2024年12月5日,中国科学院、复旦大学生命科学学院/人类表型组研究院汪思佳团队等研究人员在《国际法医学杂志》(International Journal of Legal Medicine)发表了题为“Development of a 10-CpG DNA methylation age prediction model for forensic individual identification”的研究论文。该研究基于中国汉族人群的大规模样本,构建了一个基于10个CpG位点的DNA甲基化年龄预测模型,显著提升了法医鉴定中个体年龄估计的准确性,为复杂案件侦破和未知身份识别提供了强有力的科技支撑。

随着DNA甲基化研究在法医学中的应用日益广泛,如何从个体遗留的生物样本中推断年龄成为热点课题。传统方法多依赖于少量候选位点,预测精度有限,且常面临群体适应性差与数据冗余问题。尤其是在亚洲群体中,既往研究样本量小,年龄跨度窄,尚难形成具广泛适用性的标准模型。为解决这一瓶颈,研究团队依托“国家体质调查队列”(NSPT)中来自中国汉族的3312名个体,采用Illumina 850K高通量甲基化芯片,完成了迄今为止规模最大的年龄相关全基因组甲基化关联研究。研究创新性地引入逐步条件筛选方法(SCEWAS),通过多轮迭代控制已知显著位点的共线性效应,精准识别出28个具有独立预测能力的CpG位点。
在权衡预测性能与实际应用可行性基础上,研究最终从28个位点中进一步筛选出10个信息量最丰富的CpG位点,建立了线性回归模型。该模型在2664名训练集样本中解释了93%的年龄变异(R² = 0.93),在完全独立的648人测试集中亦维持高精度(R² = 0.85,平均绝对误差MAD = 3.20岁)。此外,该模型在公开数据库中的异源群体(欧洲人、非洲人和亚洲人)中亦表现出稳健性,尽管准确率略有下降,但仍优于多数既有模型。
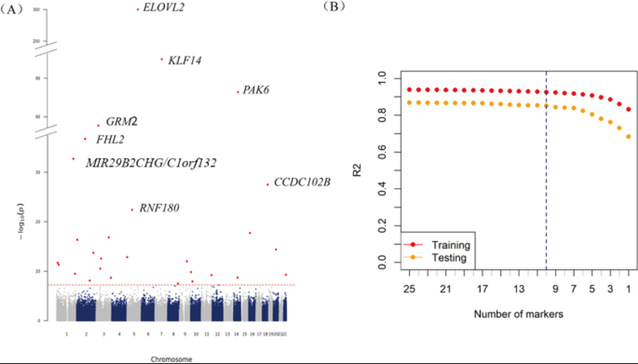
图1(A):年龄条件性全基因组甲基化关联的曼哈顿图
图1(B):后向逐步回归(BSR)中的特征选择结果
研究还比较了该模型与当前在中国男性群体中表现最优的9-CpG预测模型。在统一平台与样本条件下,10-CpG模型在预测精度(R²)与误差控制(MAD)方面均优于前者,展示出更强的泛化能力。研究同时探讨了不同建模方法,包括支持向量机(SVM)和人工神经网络(ANN),结果表明传统线性模型在解释力与应用简便性之间具备最优权衡。
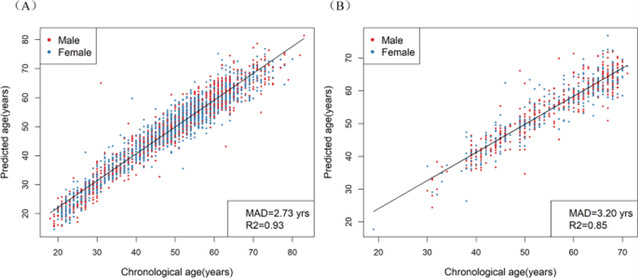
图2(A):预测年龄与实际年龄的散点图(n = 2664)
图2(B):预测年龄与实际年龄的散点图(n = 648)
值得关注的是,这10个位点分别位于ELOVL2、KLF14、FHL2、MIR29B2CHG/C1orf132、GRM2、RNF180、CCDC102B等多个基因附近,这些基因广泛参与细胞代谢、神经调控与组织发育,支撑了其在生物年龄演化过程中的作用基础。其中ELOVL2(cg16867657)为当前已知与年龄关联最强的甲基化位点,单独即可解释超过68%的年龄变异。
研究在方法论与应用层面均具重大意义。SCEWAS方法为甲基化标记的高效筛选提供了新思路,尤其适用于需兼顾精准度与样本受限的司法场景。而10-CpG模型仅依赖少量生物位点,便可在微量DNA样本中实现高精度预测,极大提升了其在现场勘查、身份识别及历史样本解析等法医应用中的可操作性。
本研究由中国科学院、复旦大学等机构合作完成,并获得国家自然科学基金、上海市科委等项目资助。
论文链接:https://link.springer.com/article/10.1007/s00414-024-03365-2
